Automatisation des flux d'analyse des flux métaboliques dans la recherche sur le cancer
Une étude comparant les cultures in vitro en 2D et 3D dans le carcinome pulmonaire non à petites cellules
En tant que chercheur spécialisé dans la métabolomique de découverte, l'un des objectifs est de mesurer les modifications du métabolome d'un système biologique. Sur la base des différents métabolites identifiés, vous pouvez alors impliquer des voies métaboliques spécifiques. Cette forme d'analyse de réseau est nécessaire pour comprendre les changements que les maladies, comme le cancer, apportent à un système biologique.
La prochaine question à laquelle il faut répondre est de savoir exactement comment ces voies intracellulaires ont changé.
L'analyse des flux métaboliques au 13C(13C-MFA) est devenue la méthode analytique de choix pour mesurer les taux de production et de consommation des voies métaboliques et, par conséquent, les changements dynamiques de ces voies.
Dès 1956, Warburg et al ont identifié la préférence de la glycolyse aérobie sur la voie de la phosphorylation oxydative dans la production d'énergie dans les cellules cancéreuses. Le 13C-MFAa enrichi ces connaissances en impliquant d'autres voies altérées et en améliorant notre compréhension du métabolisme du cancer.

Dans le cas du 13C-MFA, les données LC-MS sont souvent générées à partir de points temporels et de conditions multiples. Ces fichiers de données doivent ensuite être analysés pour déterminer l'incorporation des isotopes dans les métabolites cibles liés aux voies d'intérêt. Cette analyse nécessite de nombreuses étapes répétitives à effectuer sur plusieurs fichiers de données avant de pouvoir les interpréter. Le manque d'uniformité dans les étapes de traitement peut entraîner des erreurs, et le fait d'attendre la fin d'un processus avant de lancer le suivant ajoute un temps inutile à l'analyse.
Waters et Elucidata ont travaillé ensemble pour éliminer ces risques et ces obstacles en permettant l'automatisation des différentes étapes du traitement des données. Notre logiciel Symphony Data Pipeline peut désormais fournir à MSConvert et ElMaven les arguments de ligne de commande nécessaires pour transférer, convertir, détecter les pics et enfin télécharger les fichiers de données expérimentales vers les outils de visualisation Elucidata Polly sur elcudidata.io.
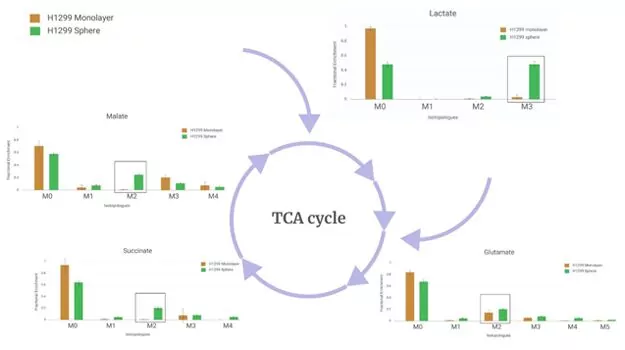
En travaillant avec nos collaborateurs de l'université de Georgetown, nous avons mis ce flux de travail à l'épreuve avec un ensemble de données modèle généré par une étude comparant une culture de cellules cancéreuses monocouche à une culture de cellules sphéroïdes. Les sphéroïdes sont populaires depuis un certain temps en raison de leur capacité à mieux représenter les micro-environnements in-vivo. Cela est particulièrement vrai dans le cas de la recherche sur le cancer et la croissance des tumeurs. L'ensemble de données devrait donc montrer une représentation assez typique des voies indicatives du métabolisme du cancer.
Vous pouvez en savoir plus sur l'expérience, les résultats et le flux de travail dans notre note d'application.
Ressources supplémentaires :
- Automatisation de l'analyse des flux métaboliques avec Symphony et Polly
- Analyse qualitative des flux de 13C avec les logiciels Symphony et Polly pour les cellules de carcinome pulmonaire non à petites cellules cultivées in vitro en deux et trois dimensions
Pour la recherche uniquement. Ne pas utiliser dans les procédures de diagnostic.
Sujets populaires
ACQUITY QDa (16) bioanalyse (11) produits biologiques (14) biopharma (26) biopharmaceutique (36) biosimilaires (11) biothérapeutique (16) étude de cas (16) chromatographie (14) intégrité des données (21) analyse alimentaire (12) HPLC (15) LC-MS (21) chromatographie liquide (LC) (19) détection de masse (15) spectrométrie de masse (MS) (54) développement de méthodes (13) STEM (12)


