Automatización de los flujos de trabajo de análisis del flujo metabólico en la investigación del cáncer
Un estudio que compara los cultivos in vitro en 2D y 3D en el carcinoma de pulmón no microcítico
Como investigador centrado en la metabolómica de descubrimiento, uno de los objetivos es medir los cambios en el metaboloma de un sistema biológico. A partir de los distintos metabolitos identificados, se pueden implicar vías metabólicas específicas. Esta forma de análisis de redes es necesaria para comprender los cambios que las enfermedades, como el cáncer, producen en un sistema biológico.
La siguiente pregunta que hay que responder es cómo han cambiado exactamente estas vías intracelulares.
El análisis del flujo metabólico con 13C(13C-MFA) se ha convertido en el método analítico de elección para medir las tasas de producción y consumo y, en consecuencia, los cambios dinámicos en las vías metabólicas.
Ya en 1956, Warburg et al identificaron la preferencia de la glucólisis aeróbica sobre la vía de la fosforilación oxidativa en la producción de energía en las células cancerosas. La 13C-MFAha ampliado estos conocimientos implicando otras vías alteradas y mejorando nuestra comprensión del metabolismo del cáncer.

En el 13C-MFA, los datos de LC-MS se generan a menudo a partir de múltiples puntos de tiempo y condiciones. Estos archivos de datos deben ser analizados para la incorporación del isótopo en los metabolitos objetivo relacionados con las vías de interés. Este análisis requiere múltiples pasos repetitivos que se llevan a cabo en múltiples archivos de datos antes de que se pueda llevar a cabo la interpretación de los datos. La incoherencia en los pasos de procesamiento puede dar lugar a errores, y esperar a que se complete un proceso antes de iniciar el siguiente añade tiempo innecesario al análisis.
Waters y Elucidata han trabajado juntos para eliminar estos riesgos y barreras permitiendo la automatización de las distintas etapas del procesamiento de datos. Nuestro software Symphony Data Pipeline ahora puede alimentar a MSConvert y ElMaven con los argumentos de línea de comandos necesarios para transferir, convertir, detectar picos y, finalmente, cargar los archivos de datos experimentales a las herramientas de visualización Elucidata Polly en elcudidata.io.
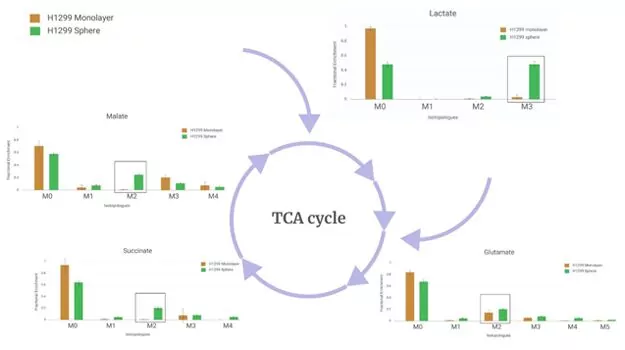
Trabajando con nuestros colaboradores de la Universidad de Georgetown, pusimos a prueba este flujo de trabajo con un conjunto de datos modelo generados a partir de un estudio que comparaba un cultivo de células cancerosas en monocapa con un cultivo de células en esferoides. Los esferoides son muy populares desde hace tiempo por su capacidad de representar mejor los microentornos in vivo. Esto es especialmente cierto en el caso de la investigación del cáncer y el crecimiento de los tumores. Por tanto, el conjunto de datos debería mostrar una representación bastante típica de las vías indicativas del metabolismo del cáncer.
Puede leer más sobre el experimento, los resultados y el flujo de trabajo en nuestra nota de aplicación.
Recursos adicionales:
- Automatización del análisis del flujo metabólico con Symphony y Polly
- Análisis de flujo cualitativo de 13C con el software Symphony y el software Polly de células de carcinoma pulmonar de células no pequeñas cultivadas in vitro en dos y tres dimensiones
Sólo para uso en investigación. No debe utilizarse en procedimientos de diagnóstico.
Temas populares
ACQUITY QDa (16) bioanálisis (11) biológicos (14) biofarmacia (26) biofarmacéutica (36 ) biosimilares (11) bioterapéutica (16) estudio de caso (16) cromatografía (14) integridad de los datos (21) análisis de alimentos (12) HPLC (15) LC-MS (21 ) cromatografía líquida (LC) (19) detección de masas (15) espectrometría de masas (EM) (54 ) desarrollo de métodos (13) STEM (12)


